통계공부합니다
[R] Kaplan-Meier Estimation with weights (가중치를 부여한 카플란 마이어 추정) 본문
[R] Kaplan-Meier Estimation with weights (가중치를 부여한 카플란 마이어 추정)
justdoit ok? 2024. 11. 15. 13:57지난 포스팅에서 R에서 IPTW와 생존분석을 진행하는 방법에 대해 알아보았다.
2024.11.09 - [Programming/R] - [R] IPTW, Inverse Probability of Treatment Weighting (역확률가중치)
2024.11.13 - [Programming/R] - [R] Survival Analysis (Kaplan-Meier, Log-rank, Cox PH)
이번 포스팅에서는 R에서 IPTW 가중치를 부여하여 Kaplan-Meier 생존 곡선을 그리는 방법에 대해 알아보겠다.
{survival, survminer} 패키지의 survfit, ggsurvplot 함수를 조합하여 그리는 방법과
{survey, jskm} 패키지의 svykm, svyjskm함수를 조합하여 그리는 방법이 있다.
1. {survival, survminer}
일반적으로 카플란 마이어 생존곡선을 그리는 방법과 유사하다.
먼저 survival 패키지의 survfit 함수로 생존 곡선을 추정해준다. 이 때, weights 인자에 IPTW 가중치를 지정해주면 가중치가 부여된 생존 곡선으로 추정할 수 있다.
iptwOS <- survfit(Surv(ET_Death,Death) ~ Group, df.surv, weights = Wate, robust = T)
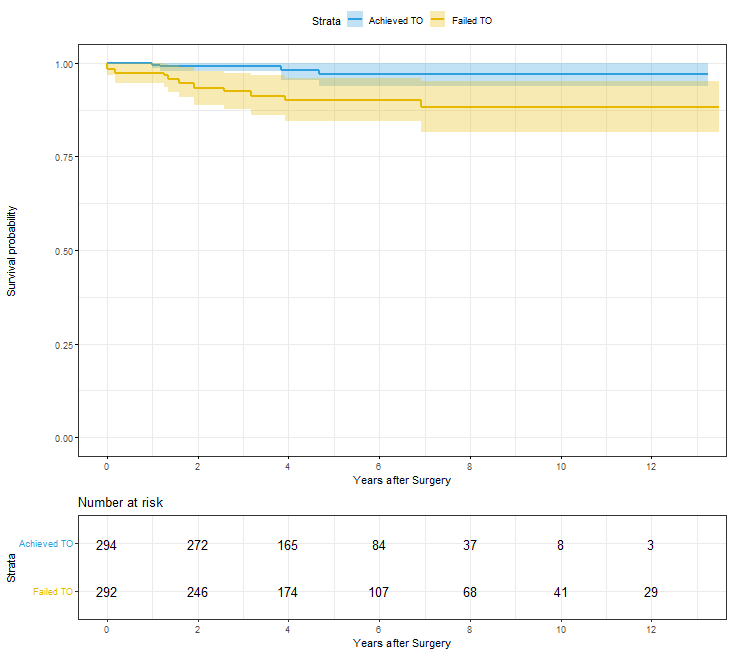
그 다음 survminer 패키지의 ggsurvplot 함수로 그래프를 그려준다.
주의해야할 점은 ggsurvplot에서 구해지는 p-value는 가중치가 부여되지 않은 데이터에서의 log-rank test 결과라는 점이다.
ggsurvplot(
iptwOS,data = df.surv,size = 1,
palette = c("#2E9FDF","#E7B800"),
censor = F, conf.int = TRUE, # 신뢰구간 표시
risk.table = TRUE, pval = F, # p-value는 False
break.x.by = 2, xlab = "Years after Surgery", xlim = c(0,10),
legend.labs = c('Achieved TO',"Failed TO"), risk.table.height = 0.25,
ggtheme = theme_bw(),
font.x = c(16), # x축 제목 크기
font.y = c(16), # y축 제목 크기
font.tickslab = c(14), # 축 텍스트(눈금) 크기
font.legend = c(12), # 범례 텍스트 크기
font.title = c(14) # 범례 제목 크기
)
2. {svykm, svyjskm}
두 번째 방법에서는 svydesign 함수로 가중치가 부여된 데이터셋을 먼저 만들어 준다.
svydesign으로 만들어진 데이터셋을 svykm 함수의 design 인자에 지정해준다. 이 때 se = T로 지정해주면 추정 시에 신뢰구간을 함께 계산하는데, 문제는 Kaplan-Meier 추정법이 아닌 Aalen (hazard-based) 추정법을 사용하게 된다.
- svykm (se=T) : Aalen (hazard-based) estimator
- svykm (se=F) : Kaplan-Meier estimator
Kaplan-Meier로 추정된 신뢰구간을 사용해야 한다면 survfit, ggsurvplot을 사용하자.
# model
iptwsvy <- svydesign(ids = ~ 1, data = df.surv, weights = ~ Wate)
iptwOS<-svykm(Surv(ET_Death,Death) ~ Group, design=iptwsvy, data=df.surv, se=F)
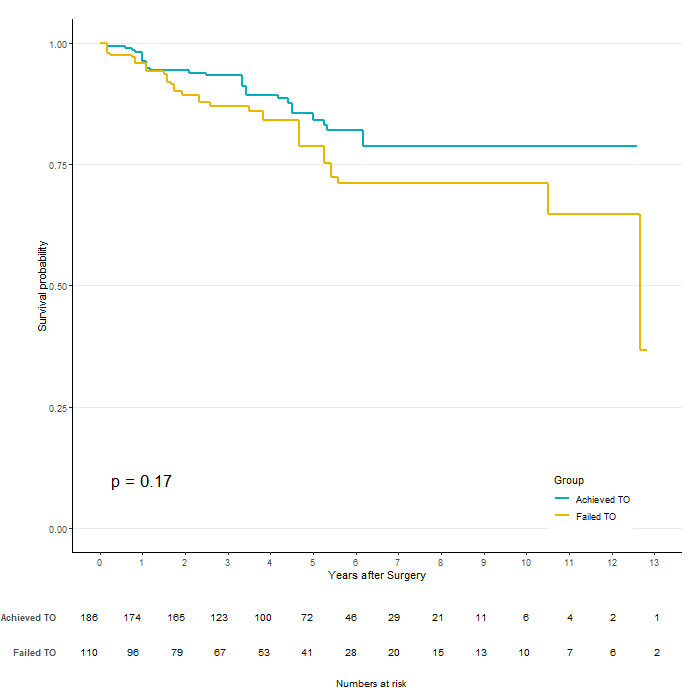
svykm 함수로 생존 곡선을 추정하였다면, svyjskm 함수로 그래프를 그려주면 된다.
여기서 나오는 p-value는 가중치가 부여된 데이터에서 log-rank test를 수행한 결과이다.
svyjskm(iptwOS, timeby = 1, ystratalabs=c('Achieved TO',"Failed TO"),
ystrataname = "Group", table = T,
pval=T, pval.coord = c(1, 0.1), pval.size = 6,
xlabs="Years after Surgery", main ="",
dashed=FALSE, marks=F, xlims=c(0,13), legendposition=c(0.85,0.1), theme = 'jama')
'Programming > R' 카테고리의 다른 글
| [R] Fleiss' kappa (플레이스의 카파) (0) | 2024.11.26 |
|---|---|
| [R] Survival Analysis (Kaplan-Meier, Log-rank, Cox PH) (0) | 2024.11.13 |
| [R] PSM, Propensity Score Matching (성향점수매칭) (1) | 2024.11.12 |
| [R] IPTW, Inverse Probability of Treatment Weighting (역확률가중치) (2) | 2024.11.09 |
| [R] R에서 변수 동적 처리 (0) | 2024.07.12 |


