통계공부합니다
[R] IPTW, Inverse Probability of Treatment Weighting (역확률가중치) 본문
[R] IPTW, Inverse Probability of Treatment Weighting (역확률가중치)
justdoit ok? 2024. 11. 9. 20:29이번 포스팅에서는 R에서 IPTW(역확률 가중치)를 수행하는 방법에 대해 알아보겠다.
이론적인 설명은 아래 포스팅을 참고하자!
[통계] IPTW, Inverse Probability of Treatment Weighting (역확률가중치)
IPTW, Inverse Probability of Treatment Weighting (역확률가중치) 성향점수분석 기법 중 하나인 처치역확률가중치(IPTW, Inverse Probability of Treatement Weighting)는 가중치를 부여하여 혼란 변수를 보정하는 기법
meowstudylog.tistory.com
예시로 사용할 데이터의 원인 변수는 치료 여부 (Group)이고 결과 변수는 사망 여부 (Death)이다.
나이(Age), 성별(Gender), 체표면적(BSA), 흡연력(Smoking), 고혈압(HTN), 당뇨(DM)가 치료-사망 간의 관계를 규명하는데 혼란 변수로 작용하는지 확인하고, IPTW를 통해 이를 보정하는 과정을 아래 절차대로 알아보자.
1. 공변량 선정
2. 성향 점수 추정
3. 공통지지영역 점검
4. IPTW 계산
5. 공변량 균형성 점검
1. 공변량 선정
먼저 공변량 선정을 위해 원인 변수 또는 결과 변수와 관련된 변수를 확인한다.
상관 분석이나 회귀 분석 등을 수행하여 관련 정도를 확인하면 되고, 나는 단변량 로지스틱 회귀분석을 통해 상관 정도를 확인했다.
## 1) 공변량 선정
# 단변량 로지스틱을 한번에 수행하기 위한 루프문
uni_logistic_tb <- function(data, y){...}
# 원인변수를 종속변수로 단변량 로지스틱 결과 확인
uni_logistic_tb(df, 'Group')
# 결과변수를 종속변수로 단변량 로지스틱 결과 확인
uni_logistic_tb(df, 'Death')
* uni_logistic_tb() 코드에 대한 내용는 여기서 확인!
> uni_logistic_tb(df.group, 'Group')
y label OR (CI 95%) p.value
2 Group Age 1.025 (1.001-1.050) 0.045
4 Group Gender1 1.156 (0.613-2.142) 0.649
6 Group BSA 0.227 (0.053-0.939) 0.043
8 Group Smoking1 1.454 (0.874-2.412) 0.147
10 Group HTN1 1.706 (0.997-2.988) 0.056
12 Group DM1 1.017 (0.634-1.633) 0.945> uni_logistic_tb(df.death, 'Death')
y label OR (CI 95%) p.value
2 Death Age 1.005 (0.959-1.056) 0.845
4 Death Gender1 1.559 (0.424-4.633) 0.455
6 Death BSA 0.148 (0.008-2.707) 0.197
8 Death Smoking1 1.653 (0.583-4.452) 0.324
10 Death HTN1 1.307 (0.447-4.747) 0.649
12 Death DM1 4.375 (1.391-19.280) 0.023
결과를 보면 원인 변수(Group)과 관련 있는 변수는 Age, BSA, HTN 임을 확인할 수 있고,
결과 변수(Death)와 관련 있는 변수는 DM 임을 확인할 수 있다.
따라서, 성향 점수 추정에 사용될 공변량은 총 4개 (Age, BSA, HTN, DM)으로 선정한다.
2. 성향 점수 추정
로지스틱 모형 등의 분류 모델을 사용하여 치료 그룹에 배치될 확률을 의미하는 성향 점수를 추정한다.
아래는 원인 변수(Group)를 종속 변수로, 공변량 4개(Age, BSA, HTN, DM)를 독립변수로 하는 로지스틱 모형을 구축하고 성향점수를 추정해준다.
## 2) 성향점수 추정
# 로지스틱 모형 적합
md.ps <- glm(Group ~ Age + BSA + HTN + DM, data = df.ps, family = 'binomial')
summary(md.ps)
# 성향점수 계산
df.ps$ps <- predict(md.ps, df.ps, type = 'response')
# 성향점수 분포 확인
plot_prob = df.ps %>% ggplot(aes(x=ps_prob))+
geom_histogram(bins = 40)+
labs(x='추정확률 형태 성향점수', y='빈도')+
theme_bw()
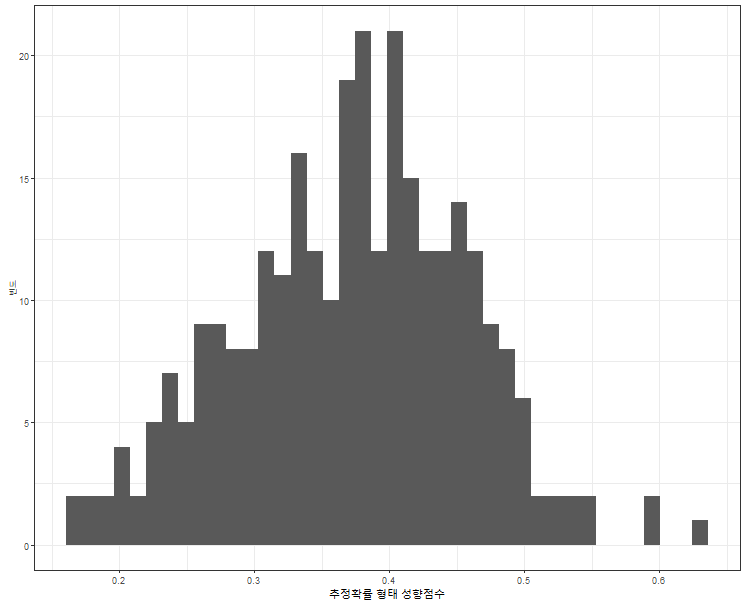
성향 점수가 고르게 분포함을 확인할 수 있다.
3. 공통지지영역 점검
추정한 성향 점수의 분포를 통제 그룹과 치료 그룹으로 구분하여 두 그룹의 성향 점수가 겹치는 영역인 공통지지영역을 확인한다.
## 3) 공통지지영역 점검
# histogram
plot_prob_cs = ggplot(data = df.ps, aes(x=ps_prob))+
geom_histogram(data=df.ps %>% filter(Group==1),
bins=40, fill='red',alpha=0.2)+
geom_histogram(data=df.ps %>% filter(Group==0),
bins=40, fill='blue',alpha=0.2)+
labs(x='추정확률 형태 성향점수', y='빈도')+
theme_bw()+
ggtitle("추정확률 이용시 공통지지영역")
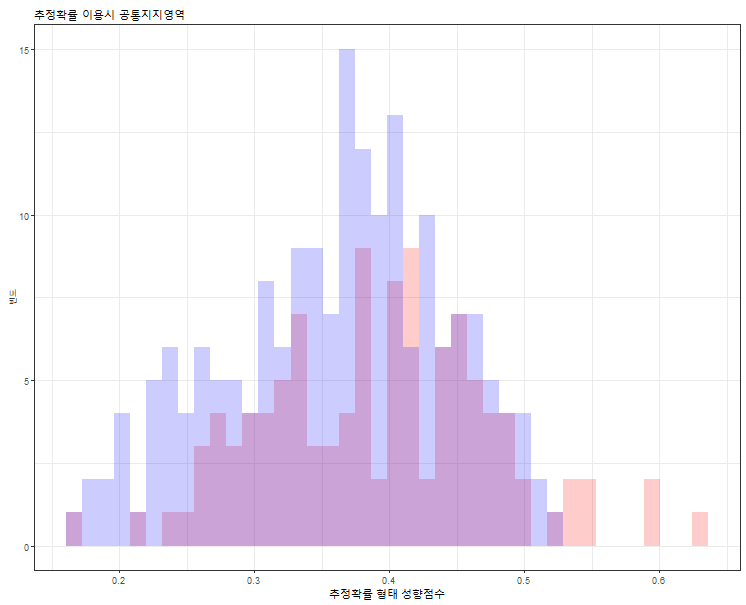
대부분의 사례들이 공통지지영역에 속해 있는 것을 확인할 수 있다.
4. IPTW 계산
추정한 성향 점수를 바탕으로 ATE, ATT, ATC를 계산한다.
만약 계산한 가중치에 극단치가 존재한다면 99%지점의 값으로 조정해줄 수 있다.
## 4) IPTW 계산
library(Hmisc)
library(twang)
# ATE, ATT, ATC 계산
df.ps <- df.ps %>%
mutate(Wate = ifelse(df.ps$Group == 1, 1 / df.ps$ps_prob, 1 / (1 - df.ps$ps_prob)),
Watt = ifelse(df.ps$Group == 1, 1 , 1 / (1 - df.ps$ps_prob)),
Watc = ifelse(df.ps$Group == 1, 1 / df.ps$ps_prob, 1)
)
# 극단치 조정 99% 지점으로
df.ps$WateTrun = ifelse(df.ps$Wate > quantile(df.ps$Wate, prob=0.99),
quantile(df.ps$Wate, prob = 0.99),
df.ps$Wate)
5. 공변량 균형성 점검
IPTW 가중치를 계산하였다면 가중치 조정 전과 후의 SMD를 비교하여 공변량의 균형이 적절히 조정되었는지를 확인한다.
가중치를 고려한 데이터 구조를 만들기 위해서는 survey 패키지의 svydesign() 함수를 사용한다.
library(tableone)
library(survey)
library(ggplot2)
library(plotly)
## 5) 공변량 균형성 점검
# 가중치 조정 전 데이터의 SMD 확인
covariates = c('Age', 'BSA','HTN','DM')
Unadjusted <- CreateTableOne(data = df.ps, vars = covariates, strata = 'Group')
ExtractSmd(Unadjusted)
# 가중치를 부여한 데이터 생성 후 SMD 확인
iptwsvy <- svydesign(ids = ~ 1, data = df.ps, weights = ~ Wate)
iptw <- svyCreateTableOne(vars = covariates, strata = 'Group', data = iptwsvy)
ExtractSmd(iptw)
# SMD 그래프 그리기
dataPlot <- data.frame(variable = rownames(ExtractSmd(Unadjusted)),
Unadjusted = ExtractSmd(Unadjusted),
Weighted = ExtractSmd(iptw))
names(dataPlot)<-c("variable",'Unadjusted','Weighted')
library(reshape2)
dataPlotMelt <- melt(data = dataPlot,
id.vars = "variable",
variable.name = "method",
value.name = "SMD")
varsOrderedBySmd <- rownames(dataPlot)[order(dataPlot[,"Unadjusted"])]
dataPlotMelt$variable <- factor(dataPlotMelt$variable,
levels = varsOrderedBySmd)
dataPlotMelt$method <- factor(dataPlotMelt$method,
levels = c("Weighted","Unadjusted"))
ggplotly(ggplot(data = dataPlotMelt, mapping = aes(x = variable, y = SMD, group = method, linetype = method, color = method)) +
geom_line() +
geom_point() +
geom_hline(yintercept = 0, size = 0.3) +
geom_hline(yintercept = 0.1, size = 0.1) +
coord_flip() +
theme_bw() + theme(legend.key = element_blank())+
ggtitle(''))> ExtractSmd(Unadjusted)
1 vs 2
Age 0.238167422
BSA 0.243463611
HTN 0.236689781
DM 0.008230615
> ExtractSmd(iptw)
1 vs 2
Age 0.001442489
BSA 0.009655934
HTN 0.019398808
DM 0.006062957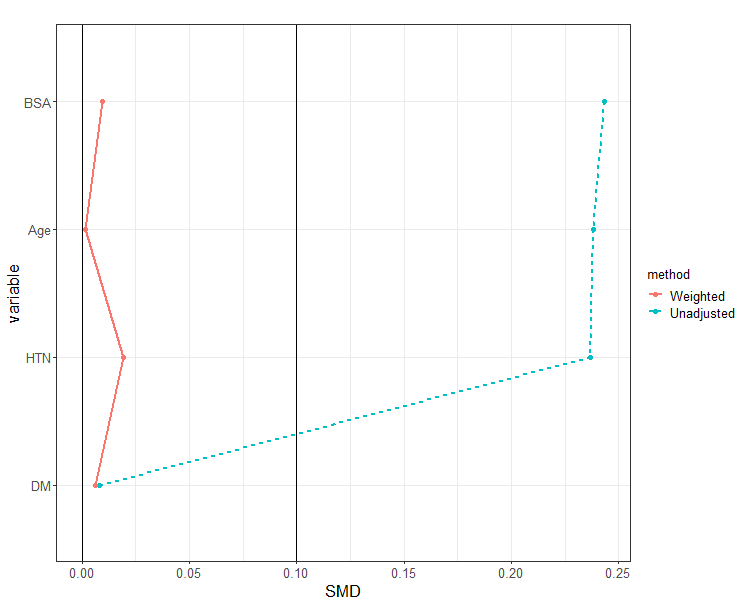
IPTW 가중치 조정 후 데이터에서 모든 공변량이 0.1이하의 SMD를 가지는 것을 확인할 수 있다.
- < 0.1: 매우 작은 효과, 두 집단이 거의 차이가 없음.
- 0.1~0.2: 작은 효과, 두 집단의 차이가 적음.
- 0.3~0.5: 중간 정도 효과, 두 집단 간 어느 정도 차이가 있음.
- > 0.5: 큰 효과, 두 집단 간 차이가 큼.
'Programming > R' 카테고리의 다른 글
| [R] Survival Analysis (Kaplan-Meier, Log-rank, Cox PH) (0) | 2024.11.13 |
|---|---|
| [R] PSM, Propensity Score Matching (성향점수매칭) (1) | 2024.11.12 |
| [R] R에서 변수 동적 처리 (0) | 2024.07.12 |
| [R] R에서 라이브러리 (패키지) 만들기 (0) | 2024.07.09 |
| [R] Caret Package로 모델 학습 및 튜닝하기 (0) | 2024.05.29 |


